La irrupción de nuevas tecnologías y el manejo de una cantidad enorme de datos han revolucionado el conocimiento en todas sus áreas. Su influencia es cada vez más determinante en la medicina, la economía, la educación, las humanidades y en cualquier otro aspecto de nuestras sociedades.
La práctica clínica del dermatólogo se ve afectada por la inevitabilidad del cambio1. La aplicación de las nuevas terapias dirigidas ha cambiado nuestros protocolos de tratamiento para algunas enfermedades como la psoriasis, la dermatitis atópica o el melanoma avanzado. Gracias a estos avances, nos hemos posicionado a la vanguardia de la inmunología, la terapia génica, la nanotecnología o la telemedicina2.
De forma paralela, el impulso tecnológico ha supuesto una explosión en la generación de datos digitales, la mejora de algoritmos y el perfeccionamiento del hardware informático3. En la era de la tecnología, la innovación exige no solo el desarrollo de nuevos fármacos, sino también de nuevas técnicas y métodos diagnósticos que mejoren la atención de nuestros pacientes1.
La aplicación de modelos matemáticos para la explicación de cuestiones médicas y biológicas es una realidad. Recientemente, la descripción de los «escutoides» como una nueva forma geométrica ha permitido explicar la arquitectura y el empaquetamiento de los epitelios curvos, abriendo el camino para entender la organización tridimensional de los órganos epiteliales4. En el ámbito de la dermatología, la geometría «fractal» se ha utilizado como un método para la evaluación de las lesiones melanocíticas. El análisis de la «lagunaridad», inicialmente descrita para caracterizar objetos geométricos irregulares cuya estructura se repite en diferentes escalas («fractales»), permitió distinguir entre un nevus y un melanoma con una sensibilidad y una especificidad del 92 y el 81%, respectivamente5.
En la era del conocimiento globalizado en que estamos entrando, todo lo que pueda ser automatizado o calculado, probablemente acabará siéndolo. Durante los últimos años, la medicina se ha estandarizado y regulado para facilitar el diagnóstico y el tratamiento de un gran número de enfermedades. Sin embargo, el uso de algoritmos en la práctica clínica potencia la aplicación de la inteligencia artificial en la toma de decisiones médicas1.
El denominado machine learning, aprendizaje automático o automatizado en español, es una forma de inteligencia artificial que tiene como objetivo desarrollar técnicas para que los ordenadores aprendan. De forma genérica, se refiere a la amplia gama de algoritmos que realizan predicciones inteligentes derivadas del análisis automático de un amplio conjunto de datos3,6. El aprendizaje automatizado es, por tanto, un concepto amplio que abarca desde formas simples de árboles de decisión o regresión lineal a complejas redes neuronales artificiales donde los datos se cuentan por millones3.
El método científico basado en la extrapolación de datos a través de muestreos aleatorios era un síntoma de nuestra incapacidad para analizar e interpretar todos los datos7. El deep learning, o aprendizaje profundo, surge como una herramienta computacional para dar respuesta a esta nueva necesidad.
Las redes neuronales artificiales, inspiradas en las células de la corteza cerebral, constituyen la base de aplicación del aprendizaje profundo8. Este entramado permite la representación de datos de alto nivel, creando millones de parámetros para la segmentación, la clasificación y la detección de imágenes3,9. En concreto, las redes neuronales convolucionales (CNN, del inglés convolutional neural network) y su propuesta de percepción multicapa han revolucionado el concepto de detección de patrones en la imagen médica3,6. Estas redes combinan múltiples capas de aprendizaje especializadas y jerarquizadas con filtros sofisticados que forman redes neuronales profundas para detectar formas complejas.
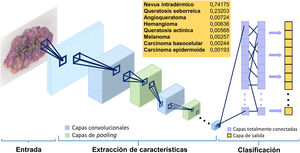
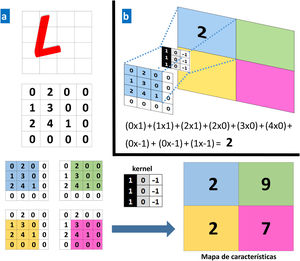
Las capas convolucionales reciben una entrada que transforman y transmiten a la siguiente. Esta modificación (operación convolucional) se realiza a través de filtros o detectores de patrones establecidos (kernel) que producen una nueva representación reducida y cada vez más abstracta de la entrada (mapas de características o feature maps). Las capas de clasificación o pooling agregan datos similares tomando la media, el máximo u otras estadísticas de los mapas de características, reduciendo así la variabilidad de los datos. Las capas finales (totalmente conectadas) clasifican los datos en una determinada etiqueta tras realizar operaciones de normalización y rectificación. De esta forma, la imagen de entrada se modela a través de sucesivas operaciones convolucionales y no lineales hasta que se transforma en una distribución de probabilidad sobre la posible pertenencia a una clase de la imagen9 (figs. 1 y 2).
Modelo de funcionamiento de una red neuronal convolucional. La imagen de entrada se transforma a través de las sucesivas capas (convolucionales y de pooling). Las capas finales (totalmente conectadas) clasifican los datos en un determinado diagnóstico11.
a. Ejemplo de una operación convolucional. Un ordenador ve una imagen (L) como una matriz de números de 4×4. Cada valor de la imagen de entrada se multiplica por un filtro o kernel. Como resultado se obtiene una nueva representación reducida (mapa de características de 2×2). b. Ejemplo de una operación concreta para los valores en el recuadro azul.
El aprendizaje profundo avanza hacia su objetivo último: la inteligencia artificial8. La mayoría de los modelos de predicción relevantes para la imagen médica se instruyen mediante colecciones de ejemplos etiquetados anteriormente (aprendizaje supervisado)3,8. Los grandes avances informáticos de los últimos años permiten a las redes neuronales asimilar un gran número de datos. Esto aumenta las conexiones entre las diferentes capas (neuronas), permitiendo que trabajen cooperativamente en la resolución de casos cada vez más complejos9. Más allá de esto, el gran avance de las redes neuronales radica en que, a diferencia de la inteligencia artificial tradicional, estas pueden elaborar de forma autónoma las normas que les permitan clasificar una determinada característica a partir de datos sin procesar3,9–11. Es decir, aprenden por sí mismas, optimizando sus predicciones y mejorando su eficacia.
Por todo esto, las CNN muestran un potencial prometedor en la imagen médica. Especialidades como la dermatología, la radiología o la anatomía patológica son el ámbito de aplicación ideal para sistemas basados en la clasificación, la segmentación y la detección de imágenes3.
En el año 2017, Esteva et al.6 describieron la aplicación de las CNN para la evaluación clínica de lesiones cutáneas. En este trabajo compararon la capacidad diagnóstica de 21 dermatólogos expertos con una CNN entrenada con 127.463 imágenes para la clasificación binaria (benigna o maligna) de lesiones de origen epidérmico y melanocítico. Los especialistas identificaron un 95% de los tumores malignos y un 76% de las lesiones benignas, mientras que la máquina realizó diagnósticos correctos en un 96 y un 90% de los casos, respectivamente. En otro trabajo, un algoritmo de fusión de aprendizaje automatizado superó el rendimiento de la mayoría de los dermatólogos en la clasificación de 100 imágenes dermatoscópicas de nevus y melanoma12.
La aplicación de las redes neuronales a la dermatología no se limita al cáncer cutáneo. DeepGestalt es un sistema diseñado para la identificación de fenotipos faciales de trastornos genéticos que combina métodos de visión e inteligencia artificial. Este programa se mostró superior a los clínicos para la detección de síndromes y podría ampliar el rendimiento de las pruebas genéticas13. Han et al. evaluaron la precisión de una CNN entrenada con 49.567 imágenes para el diagnóstico de onicomicosis. De nuevo, un sistema basado en el aprendizaje profundo superaba a la mayoría de los dermatólogos que participaron en el estudio14.
En el último año se ha producido una explosión de trabajos. El avance de estos sistemas parece imparable y, en este sentido, surgen preguntas obligadas: ¿sustituirán estos sistemas algún día la figura del dermatólogo?, ¿cómo se integrarán estos cambios en el futuro de la especialidad?15,16.
Las decisiones terapéuticas o los algoritmos diagnósticos podrán realizarse con una mayor evidencia científica. Los ordenadores, con su capacidad ilimitada de aprendizaje, sustituirán en muchos aspectos lo que hoy es competencia exclusiva del juicio humano. Los denominados big data hacen referencia a un conjunto de datos o combinación de conjunto de datos de un volumen, complejidad y velocidad de crecimiento tales que hacen imposible su procesamiento y análisis mediante tecnologías y herramientas convencionales. Su análisis nos permitirá obviar los sesgos ineludibles que genera la aleatoriedad. El éxito de la inteligencia artificial dependerá de la colaboración del dermatólogo en el desarrollo de algoritmos adecuados y grandes archivos públicos que permitan generalizar los resultados17. La tecnología del blockchain o «cadena de bloques» se extenderá más allá de sus aplicaciones financieras (Bitcoin). Esto hará posible el almacenamiento e intercambio de imágenes codificadas, ofreciendo una solución a la privacidad, la seguridad y la distribución de los datos18.
A pesar de los potenciales beneficios de la inteligencia artificial, se desconocen muchos aspectos relacionados con su aplicación19. La mayoría de estos sistemas no ha sido evaluado en un entorno clínico real. En la práctica, se requieren más criterios que el morfológico para la toma de decisiones médicas16. El manejo de un paciente es un proceso complejo y multifactorial que considera, además de datos clínicos, vías alternativas (por ejemplo, el seguimiento estrecho) a las variables dicotómicas computacionales («benigno/maligno», «extirpación/no requiere tratamiento»)3,16. Los modelos matemáticos no proporcionan un diagnóstico, sino una probabilidad diagnóstica, un valor entre 0 y 1 que en el futuro podría orientar a médicos menos experimentados o servir incluso como método de cribado, siempre como un sistema que complementa, pero no reemplaza las capacidades de un dermatólogo15,20.
Los algoritmos se incluirán en la práctica dermatológica y aumentarán su precisión diagnóstica en manos expertas12. Sin embargo, en la relación médico-paciente, la información y el conocimiento suponen solamente uno de los peldaños para ejercer una medicina de calidad. Los factores humanos seguirán teniendo un peso irremplazable por los ordenadores. Otros aspectos ineludibles en el buen hacer profesional, como la empatía y el acompañamiento del paciente, el sentido común, la creatividad, la pasión, la motivación y la innovación, harán irremplazable la figura del dermatólogo.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.