La dermatitis atópica es el trastorno inflamatorio de la piel crónico más común. Afecta hasta a 20% de los niños y a 10% de los adultos en países desarrollados. La fisiopatología de la dermatitis atópica es compleja e implica una fuerte predisposición genética e inflamación impulsada por células T. Aunque nuestra comprensión de la patología y las causas de esta enfermedad ha mejorado en los últimos años, aún existen lagunas de conocimiento en las vías inmunológicas involucradas. En consecuencia, los avances en nuevas tecnologías ómicas en la dermatitis atópica desempeñarán un papel clave en la comprensión de la patogénesis de esta enfermedad y podrían desarrollar estrategias preventivas y tratamientos personalizados. En esta revisión se discuten los últimos avances en genética, transcriptómica, epigenómica, proteómica y metagenómica, y entendemos cómo la integración de múltiples conjuntos de datos ómicos identificará posibles biomarcadores y descubrirá redes de asociaciones entre varios niveles moleculares.
Atopic dermatitis is the most common chronic inflammatory skin disorder, affecting up to 20% of children and 10% of adults in developed countries. The pathophysiology of atopic dermatitis is complex and involves a strong genetic predisposition and T-cell driven inflammation. Although our understanding of the pathology and drivers of this disease has improved in recent years, there are still knowledge gaps in the immune pathways involved. Therefore, advances in new omics technologies in atopic dermatitis will play a key role in understanding the pathogenesis of this burden disease and could develop preventive strategies and personalized treatment strategies. In this review, we discuss the latest developments in genetics, transcriptomics, epigenomics, proteomics, and metagenomics and understand how integrating multiple omics datasets will identify potential biomarkers and uncover nets of associations between several molecular levels.
La dermatitis atópica (DA) ha experimentado, durante la última década, una revolución en el conocimiento acerca de su patogenia, lo que ha permitido la utilización, por primera vez en su historia, de estrategias terapéuticas personalizadas. Sin embargo, la DA es una enfermedad compleja, muy heterogénea no sólo desde el punto de vista clínico, sino también con importantes variaciones asociadas a la edad, los grupos étnicos y la presencia o ausencia de comorbilidades relacionados con la atopia y enfermedades concomitantes. La mayoría de los conocimientos generados tiene el riesgo de no poder caracterizar de manera adecuada el amplio espectro de pacientes incluidos bajo este espectro, especialmente si estos no se integran de manera adecuada de manera que puedan ser interpretados por el dermatólogo clínico.
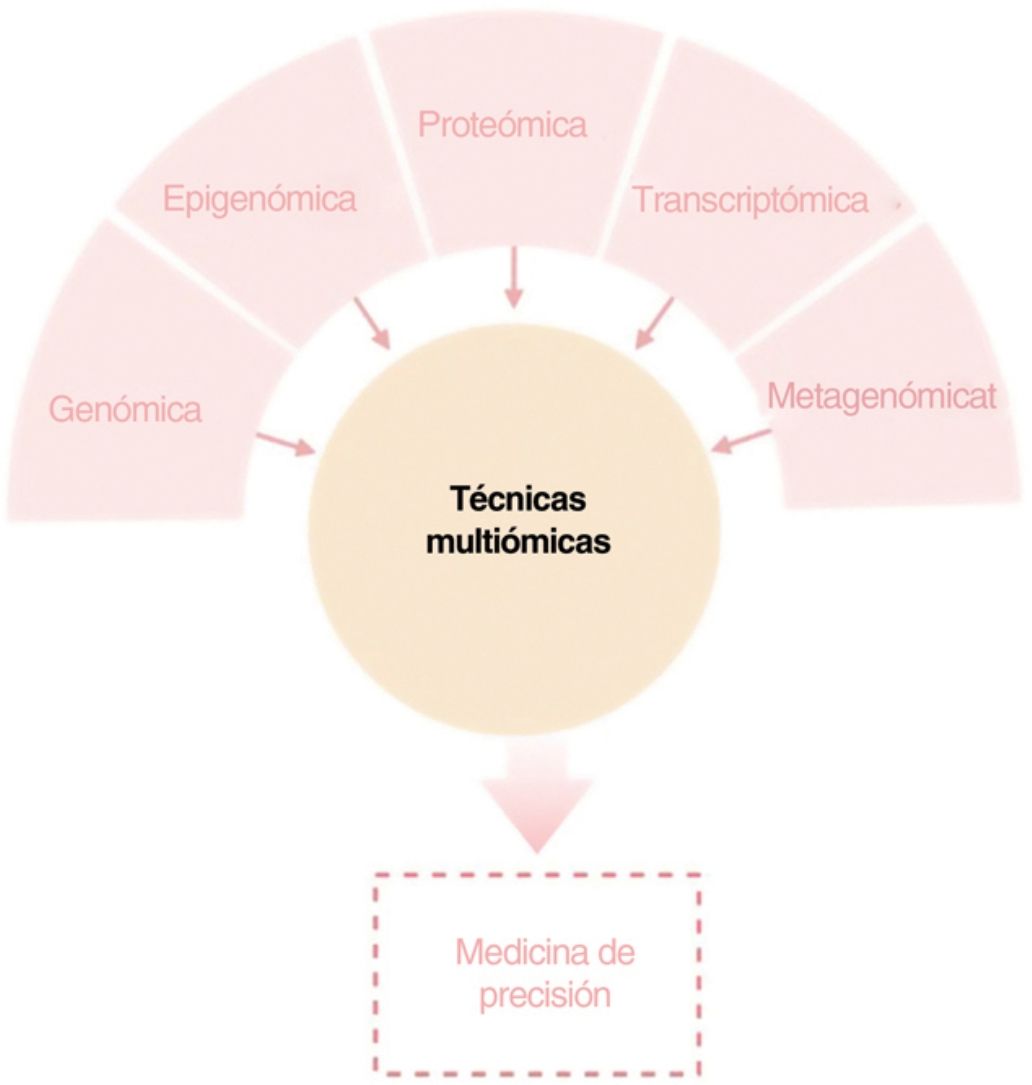
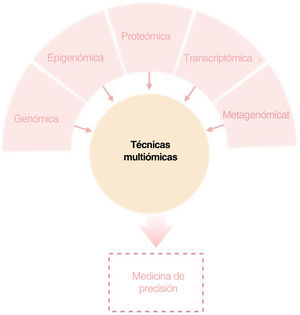
El objetivo de esta revisión es ofrecer a los dermatólogos clínicos una visión general de la naturaleza y los objetivos de las principales tecnologías ómicas (fig. 1).
GenéticaHeredabilidadLa dermatitis atópica (DA) es una enfermedad inflamatoria inmunomediada compleja que tiene un fuerte componente genético1. Se ha estimado que la heredabilidad de la DA es del 70-80%2,3, lo que demuestra el importante papel que desempeñan los factores genéticos en el desarrollo de este trastorno inflamatorio crónico. En concreto, los antecedentes familiares de la DA se han descrito como el factor de riesgo de enfermedad más potente. Hasta la fecha, los factores de riesgo genéticos subyacentes a la DA se han identificado mediante estudios de vinculación genética, enfoques de genes candidatos, estudios de asociación de todo el genoma (GWAS) y, más recientemente, las tecnologías de secuenciación de nueva generación (NGS) también han contribuido a comprender mejor la base del riesgo genético de la DA.
Estudios de ligamiento y abordaje de genes candidatosLos estudios de ligamiento genético tienen por objeto comparar la herencia de marcadores genéticos con la presencia de un rasgo clínico para identificar genes de riesgo de enfermedad. En el año 2000, el primer estudio de ligamiento genómico en la DA identificó un locus de susceptibilidad importante en el cromosoma 3q214. El abordaje de los genes candidatos, en el que se compara la variación en genes que se sospecha que están implicados en la patogénesis de la enfermedad entre individuos afectados y sanos, se ha utilizado en más de 30 estudios para identificar loci asociados al desarrollo y la gravedad de la DA5. El gen FLG, es el gen que codifica para la filagrina; es uno de los genes más conocidos asociados tanto a la susceptibilidad como a la gravedad de la enfermedad1. También se han estudiado múltiples genes que, por su implicación funcional en la biología de la enfermedad, son determinantes genéticos del riesgo de DA5. Por ejemplo, se ha descubierto que las mutaciones de pérdida de función en el gen FLG son uno de los principales impulsores del desarrollo de la DA, y que estas mutaciones provocan la pérdida de expresión de la filagrina, una proteína estructural que une las fibras de queratina en el epitelio6,7. Asimismo, estudios realizados en la población asiática han identificado una asociación significativa entre el gen SPINK5, que codifica para un inhibidor de la serina proteasa que participa en el mantenimiento de la barrera cutánea, y la susceptibilidad a la DA8. También se han detectado más de 25 factores de riesgo genéticos mediante un enfoque de genes candidatos, donde se incluyen genes de las familias de la claudina (CLDN) y los receptores tipo Toll (TLR), así como ADAM, RETN, TGFB1, CD207, TSLP, IL-13, THSD7B, IL18R1, NFATC2, PTGER4, WDR36, KIF3A o MIF, entre otros5. Con la llegada de las tecnologías de secuenciación de alto rendimiento y de nueva generación, los análisis de ligamiento y de genes candidatos han sido sustituidos por estrategias analíticas más potentes que permiten un análisis exhaustivo de la variación del riesgo genético a escala de todo el genoma.
Estudios de asociación de genoma completo (GWAS)Los GWAS son un enfoque estadístico que compara la frecuencia alélica de millones de variantes genéticas entre pacientes y sujetos sanos para identificar loci asociados a rasgos clínicos9. Hasta la fecha, se han realizado un total de siete GWAS en la DA10–16. El resumen de los resultados estadísticos obtenidos de estos GWAS provenientes de poblaciones de diferentes ascendencias han sido objeto de posteriores metanalisis17–22. En conjunto, estos estudios han identificado más de 40 genes asociados al riesgo de DA. Entre ellos se incluyen genes de riesgo bien establecidos como el de la FLG, así como otros loci de riesgo como SERPINB7, DSC1 e ITGB8, entre otros. Los análisis subsiguientes de los resultados de los GWAS también han revelado un solapamiento sustancial de los marcadores genéticos de riesgo de DA con potenciadores característicos de las células de la piel y de células inmunitarias como las células T CD4+, lo que aporta pruebas de la existencia de nuevas vías biológicas implicadas en la fisiopatología de la DA.
En los estudios de asociación de todo el fenotipo, en los que se comprueba simultáneamente la asociación de una variación genética de interés con múltiples fenotipos clínicos, se ha descubierto que una variante de pérdida de función en la FLG está asociada con diferentes fenotipos clínicos de la DA, como el asma, la rinitis alérgica y la alergia alimentaria23. La variación genética en los genes de la metaloproteasa ADAM33 y del receptor de monocitos CD14 también se ha asociado con la bronquitis alérgica y el asma, respectivamente24.
Secuenciación de nueva generaciónLa tecnología de secuenciación del exoma completo (WES, por sus siglas en inglés), que permite una identificación con una elevada fiabilidad de la variación genética de las regiones exómicas, se está utilizando cada vez más para avanzar en la caracterización de los factores genéticos subyacentes a la DA25–28. Cabe destacar que se ha descubierto que una única mutación en CARD11 causa defectos celulares potencialmente corregibles que conducen a la DA29. Asimismo, la WES realizada en pacientes con DA de la población africana ha revelado variantes raras en la FLG y varios otros genes del complejo de diferenciación epidérmica que son importantes para el desarrollo de la enfermedad, así como mutaciones “nonsense” y “missense” en genes de los que no se había informado anteriormente, como el GTF2H5, EVPL o NLRP130. Otros estudios basados también en tecnologías de secuenciación de nueva generación han contribuido a identificar mutaciones “nonsense” raras y de baja frecuencia en el gen LRRC32 en pacientes con DA31, así como mutaciones adicionales en el gen de la FLG que están asociadas al desarrollo de la DA32–34.
Puntuaciones de riesgo poligénico (del inglés, Polygenic risk scores)Desde el punto de vista clínico, existe una necesidad urgente de prevenir el desarrollo de la DA y sus comorbilidades alérgicas asociadas35. En los últimos años, se están utilizando cada vez más las puntuaciones de riesgo poligénico (PRS) para identificar a aquellos individuos que presentan un riesgo elevado de padecer DA y que, por tanto, tienen más probabilidades de beneficiarse de una intervención terapéutica temprana35-37. La puntuación de riesgo poligénico (PRS) es una medida cuantitativa que estima la carga de riesgo genético de un individuo para desarrollar una enfermedad concreta38. Esta puntuación se calcula agregando el riesgo conferido por múltiples variantes de pequeño efecto que están distribuidas por todo el genoma39. Aunque hasta ahora sólo se han desarrollado unas pocas PRS para la DA40, estas puntuaciones basadas en la genética ya han demostrado un alto poder para predecir el desarrollo de la DA, alcanzando hasta un 85% de precisión diagnóstica. Al igual que en las enfermedades cardiovasculares41, la integración de los PRS con factores de riesgo clínicos establecidos podría ayudar a los médicos a detectar a los pacientes con mayor riesgo de DA y, en consecuencia, a anticipar las intervenciones terapéuticas.
Aleatorización mendelianaDeterminar la causalidad en enfermedades como la DA es clave para desarrollar mejores terapias y comprender la heterogeneidad de los pacientes. La aleatorización mendeliana (AM) es una técnica estadística que utiliza variantes genéticas como variables instrumentales para evaluar la relación causal entre una exposición y un resultado. Con la creciente disponibilidad del GWAS, la AM ha ganado popularidad en los últimos años como método para investigar posibles relaciones causales en estudios observacionales. Al utilizar variantes genéticas como sustitutos del factor de riesgo de interés, la AM minimiza el riesgo de confusión y causalidad inversa, y proporciona una estimación más sólida de la causalidad. La AM se ha aplicado a diversas áreas de investigación, incluido el estudio de las causas y consecuencias de la DA. En particular, la AM ha aportado nuevas y valiosas relaciones causales entre la DA y rasgos epidemiológicos42, clínicos43 y moleculares44.
EpigenómicaLa prevalencia de la DA ha aumentado con demasiada rapidez para que pueda explicarse por cambios en la variación genética. Se cree que estos cambios se deben a factores ambientales, como el estilo de vida, la dieta y la contaminación, mediados por el control epigenético de la expresión génica. El epigenoma puede dividirse en tres grupos principales: Metilación del ADN, modificaciones de las histonas y ARN no codificantes.
La metilación del ADN se produce cuando las metiltransferasas del ADN (DNMT) catalizan la adición de un grupo metilo al quinto carbono de la citosina, dando lugar a la 5-metilcitosina (5mC), y la presencia de este grupo metilo suele correlacionarse con la represión de la transcripción45. Varios estudios han tratado de investigar los cambios en la metilación del ADN en la DA con el objetivo de determinar un impacto mecanicista en la expresión génica patogénica. Uno de los primeros estudios que examinó las diferencias en la metilación del ADN entre la DA y los controles sanos descubrió que, aunque no había cambios en la metilación del ADN en las poblaciones de células inmunitarias, sí había alteraciones sorprendentes en la metilación del ADN en la epidermis lesional de la DA en comparación con los controles sanos. Estos cambios se correlacionaban en parte con alteraciones en la expresión de genes que participaban en la diferenciación de los queratinocitos y en la respuesta inmunitaria innata46. En un estudio más reciente, los autores observaron metilación específica del ADN celular en células T circulantes de pacientes con DA47. Más concretamente, se observó que las células T CD4+ CLA+ “homing” de la piel de pacientes con DA regulaban incrementando la expresión de la IL13, un conocido marcador Th2 de la DA, debido a la reducción de la metilación del ADN regulando al incremento de ese gen. Otros estudios identificaron alteraciones en la metilación del ADN en genes patogénicos clave, como la hipermetilación del promotor de NLRP2 en células sanguíneas de pacientes pediátricos con DA41,48, la desmetilación mediada por SNP del promotor de VSTM1 en piel lesionada por DA49, y la hipermetilación, y por tanto la regulación disminuyendo, el gen KIF3A, que codifica una proteína estructural50.
Las modificaciones de las histonas son reguladores epigenéticos clave que controlan la estructura de la cromatina y, por tanto, los eventos transcripcionales. El papel de las modificaciones de las histonas no se ha explorado bien en la DA. No obstante, se han descrito alteraciones de las modificaciones de las histonas en contextos inflamatorios relacionados; por ejemplo, un estudio describió un aumento de las marcas de histonas H3ac y H4ac transcripcionalmente activas en células T CD4+ de pacientes con asma que se correlacionaba con un aumento de la expresión de IL1351. Un estudio más reciente demostró que el ácido butírico aislado en el Staphylococcus epidermidis, una bacteria patógena conocida en la DA, funciona como inhibidor de la histona desacetilasa e induce la acetilación de H3K9 en queratinocitos humanos, lo que se correlaciona con una disminución de la producción de la IL652. Curiosamente, el butirato derivado del microbioma ha demostrado proporcionar funciones protectoras en el tejido intestinal, lo que indica un efecto altamente contextual de los metabolitos en la respuesta de las células inmunitarias.
Los ARN no codificantes son ARN funcionales que no se traducen en proteínas. Algunos de estos ARN, como los microARN (miARN), controlan directamente la expresión génica mediante la inhibición postraduccional de los transcritos de ARNm y su degradación. En la DA, se ha observado que un miARN, miR-335, está regulado de manera decreciente en las lesiones cutáneas afectadas en comparación con los controles sanos, y que su diana SOX6 está regulando hacia el incremento de forma aberrante53. Otro miARN clave que se ha descrito que está alterado en la DA es el miR-124, que está regulado a la disminución en la piel lesional de la DA en comparación con la piel no lesional. Los autores descubrieron que la expresión de miR-124 es esencial para la homeostasis de la piel al regular la actividad de p65, y que su regulación decreciente en la DA provoca una inflamación mediada por p6554.
ProteómicaEs un hecho aceptado que, aunque el análisis transcriptómico puede ser muy esclarecedor, el control de la expresión proteica puede ser independiente de la expresión génica. De ahí que los estudios proteómicos puedan ser clave para determinar los mecanismos que impulsan la patogénesis de una enfermedad. Los métodos convencionales para estudiar las proteínas incluyen la realización de ELISA y de western blot, y el SDS-PAGE y la electroforesis en gel 2D y técnicas relacionadas se utilizan para la separación de muestras de proteínas complejas55. Posteriormente, los métodos basados en la espectrometría de masas (EM) han dominado el campo de la proteómica de alto rendimiento y se han utilizado ampliamente para estudiar numerosos contextos de enfermedades. Desde entonces, se han desarrollado otros métodos de “altoplex” para estudiar la proteómica, incluidos los “arrays” de anticuerpos/antígenos56, los ensayos basados en aptámeros57 y el ensayo de extensión de proximidad (PEA)58, y estos métodos han sido clave para comprender los mecanismos de las enfermedades y guiar el descubrimiento de biomarcadores.
Entre las proteínas que inicialmente se descubrió que estaban implicadas en la patogénesis de la DA mediante métodos proteómicos convencionales realizados en piel afectada se encontraban las proteínas funcionales de unión al calcio S100/A11 y CLIC4, y las proteínas estructurales vinculina y filamina A59–61. Estudios posteriores realizados mediante cromatografía líquida-espectrometría de masas (LC-MS) revelaron reducciones significativas en los niveles de proteínas relacionadas con la generación de humedad natural y la función de barrera, lo que sugiere que el ciclo continuo de piel seca puede predisponer a los pacientes con DA a las infecciones62,63.
En un estudio de Pavel et al64, los autores observaron marcadores proteicos inflamatorios elevados en la piel lesional de la DA que incluían proteínas implicadas en la activación de células inmunitarias (TNFSF10, IL2RA y CD40) y proteínas relacionadas con Th2-, Th1- y Th17/Th22. Curiosamente, estas alteraciones también se observaron en biopsias de piel no lesional de pacientes con DA. Además, muchos de los cambios detectados a nivel proteico no se detectaron a nivel de expresión génica, lo que apoya la necesidad de realizar estudios proteómicos. Los autores también investigaron la proteómica del suero emparejado de los mismos pacientes y descubrieron que se podían detectar pocos cambios proteómicos en el suero. Entre las alteraciones detectadas, la mayoría coincidían con las detectadas en la piel, y éstas incluían proteínas asociadas a vías inmunitarias relacionadas con Th1-, Th2- y Th1764.
Otros estudios proteómicos en suero no sólo demostraron un aumento de los mediadores de la inflamación sistémica, sino también un aumento de los marcadores de riesgo cardiovascular (MMP10, SELE) y aterosclerosis (CCL4, SORT1)65,66, lo que indica su relevancia para estudiar biomarcadores que indican afecciones inflamatorias comórbidas.
Por último, se han utilizado métodos proteómicos para evaluar la respuesta de los pacientes al tratamiento. Thijs y sus compañeros analizaron los niveles de citocinas en muestras de suero de pacientes antes y después del tratamiento con corticosteroides tópicos potentes, y observaron una disminución de varias quimiocinas y citocinas, como la CCL17, la CCL18, la IL22 y la IL16, así como la IL2R y la E-selectina67. Otro estudio demostró que los niveles séricos de la IL16 se correlacionaban positivamente con la gravedad de la enfermedad y disminuían con la mejoría clínica tras el tratamiento tópico68. Un reciente ensayo clínico aleatorizado con el inhibidor de JAK/SYK ASN002 demostró que los pacientes tratados reducían los marcadores de proteínas inflamatorias sistémicas, así como los marcadores de riesgo de aterosclerosis69.
Estos resultados indican que la DA es una verdadera afección inflamatoria sistémica y que el proteoma circulante es un reflejo directo de la inflamación cutánea local, por lo que debería ser usado en el hallazgo de biomarcadores.
TranscriptómicaSe han realizado amplios estudios de transcriptómica en la DA con el objetivo de descifrar su fisiopatología. Está bien establecido que se producen cambios globales en el escenario transcriptómico de la DA en el lugar de la inflamación, más concretamente en la epidermis y la dermis, impulsados principalmente por los queratinocitos y fibroblastos activados y las células inmunitarias infiltrantes.
En primer lugar, varias proteínas estructurales están desreguladas en la DA, lo que provoca importantes alteraciones en la barrera cutánea. Desde entonces, también se ha descrito la expresión aberrante de otras proteínas estructurales, incluidas las proteínas implicadas en la formación de uniones estrechas, como varios miembros de la familia de las claudinas70, y la producción de colágeno, como COL6A5 y COL6A671. En segundo lugar, aparte de los genes de barrera, los queratinocitos de la DA desregulan muchos genes en el entorno lesional que perpetúan la patogénesis. Se ha observado que los queratinocitos de la DA regulan incrementando las proteasas, como las calicreínas y las serpinas, y disminuyendo los inhibidores de proteasas71. Además, también se alteran los genes relacionados con el metabolismo lipídico y las funciones metabólicas72. Y lo que es más importante, estos queratinocitos patógenos regulan incrementando una serie de citocinas y quimiocinas, como la CCL17, la CCL18, la CCL22 y la IL36G, que desempeñan papeles importantes en las respuestas inflamatorias mediadas por Th2- y Th22- y Th1770,71. Por último, en los estudios transcriptómicos de la piel lesional de la DA se han observado alteraciones importantes en las poblaciones de células inmunitarias cutáneas. Los genes desregulados incluyen mediadores clave de la activación de células T, como la IL22, el TNFSF4 y el CTLA4, la actividad citotóxica, el GZMB, y genes asociados con el tráfico de células T, incluido el receptor de “homing” de órganos linfoides CCR770.
Aunque los datos transcriptómicos de la piel son muy informativos en cuanto a la caracterización general de la patología de la DA, existen pruebas sólidas que sugieren la existencia de un componente sistémico que impulsa los mecanismos de la enfermedad. Un estudio realizó análisis transcriptómicos en muestras de sangre total de pacientes pediátricos con DA y observó que los marcadores de eosinófilos y Th2, incluidos la IL5RA, la IL1RL1, la CCR3, la IL13 y la IL4, estaban regulados incrementándose, mientras que los marcadores Th1, como el IFNG y el TNFA, estaban regulados disminuyendo73. Un estudio más reciente corroboró estos hallazgos en pacientes adultos con DA74. Además, los autores pudieron identificar dos endotipos de DA transcripcionalmente distintos, anotados como eosinófilo-alto y eosinófilo-bajo, caracterizados por la expresión diferencial de la firma relacionada con los eosinófilos. El genotipo de eosinófilos altos mostró una desregulación global más pronunciada y una alteración continua de la función de las células NK a pesar de la mejoría clínica tras el tratamiento. Y lo que es más interesante, el endotipo de eosinófilos bajos corresponde a una mayor proporción de super respondedores al dupilumab, un tratamiento anti-IL4RA74.
En general, las principales alteraciones transcriptómicas impulsan la inflamación cutánea local en las lesiones de DA. Sin embargo, la comprensión de los cambios transcriptómicos sistémicos en las células inmunitarias circulantes parece ser clave tanto en la búsqueda de nuevos tratamientos terapéuticos como en la determinación de la respuesta al tratamiento.
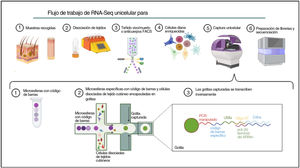
Ómicas unicelulares (del inglés Single-cell omics)El contenido celular de la piel humana se ha estudiado tradicionalmente mediante estrategias que combinan la clasificación celular activada por fluorescencia (FACS) y la inmunohistoquímica (IHC) en tejido digerido enzimáticamente. Con la reciente implementación de enfoques ómicos unicelulares, los investigadores pueden llevar a cabo una caracterización molecular y funcional exhaustiva de las células de la piel para comprender las bases moleculares de la DA75. Entre todas las tecnologías unicelulares disponibles, el protocolo basado en microfluidos en gotas comercializado por 10X Genomics es el más popular para estudiar el transcriptoma de células individuales (scRNA-Seq) en la investigación dermatológica. Esta técnica se basa en la encapsulación de células en microgotas con códigos de barras celulares e identificadores moleculares únicos (fig. 2).
El aislamiento de células individuales a partir de biopsias de piel requiere un paso inicial de tratamiento mecánico o enzimático, que puede resultar bastante difícil debido a la complejidad estructural y la heterogeneidad de la piel. Por lo tanto, el método de disociación utilizado para la obtención de células aisladas debe adaptarse a las propiedades de la capa cutánea de interés76.
La comparación de la piel de pacientes con DA y voluntarios sanos mediante scRNA-Seq ha revelado la expansión de la ILC2, células T tipo2/tipo 22, DC inflamatorias y fibroblastos que expresan citocinas proinflamatorias, como CCL2 y CCL19, en muestras de DA lesional77–79. En ensayos preclínicos, se ha demostrado que la expresión de CCL11 en una población específica de fibroblastos de sujetos con DA contribuye a la patogénesis a través de la señalización IKKB/NF-kB80. El estudio de los perfiles transcriptómicos y proteómicos de células cutáneas procedentes de la DA espontáneamente resuelta, DA crónica y donantes sanos reveló grandes diferencias reguladoras tanto en células inmunes como no inmunes entre las muestras analizadas81. También se descubrió que los melanocitos, más que las células inmunes, muestran el mayor número de genes expresados diferencialmente entre la DA espontáneamente resuelta y los controles. Este inesperado papel inmunológico de este tipo celular en la DA demuestra claramente el poder de los enfoques unicelulares para generar nuevas propuestas patogénicas de la enfermedad.
Un estudio reciente en el que participaron diferentes enfermedades cutáneas informó de la desregulación transcripcional de las células T de memoria residentes en la piel en la DA y la psoriasis. Es probable que este programa de células T contribuya de forma decisiva a la recurrencia de la enfermedad82. En la DA se observaron exclusivamente anomalías reguladoras relacionadas con los marcadores T helper en las ILC y las células T citotóxicas CD8+. La caracterización de estos cambios transcripcionales permitió definir las principales clases de la enfermedad asociadas a la respuesta terapéutica.
En resumen, los estudios de composición celular y rasgos transcripcionales de la DA realizados en los últimos cuatro años, han corroborado el papel de las células inmunes y los fibroblastos en el desarrollo de la DA, y la relevancia de los melanocitos en el mantenimiento del microambiente regulador en la remisión de la DA.
MetagenómicaLa piel humana está colonizada durante el periodo postnatal por microorganismos que impiden la invasión de patógenos externos. Este conjunto de comensales, que incluye bacterias, hongos y virus, se conoce como microbiota cutánea. La interacción entre la microbiota cutánea y el sistema inmunitario del huésped es necesaria para desencadenar la activación homeostática de los sistemas inmunitarios innato y adaptativo83.
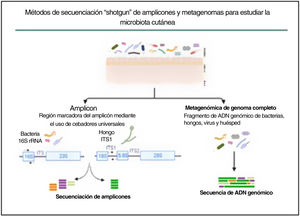
En la última década se han explorado comunidades microbianas complejas, como la microbiota de la piel humana, utilizando la metagenómica, que elude el sesgo impuesto por el cultivo bacteriano tradicional en condiciones de crecimiento artificiales. Se han desarrollado dos enfoques metagenómicos diferentes para determinar y cuantificar la composición de las comunidades microbianas: la secuenciación de amplicones y la secuenciación del metagenoma completo (fig. 3).
Métodos de secuenciación en escopeta de amplicones y metagenomas para estudiar la microbiota cutánea. La estrategia basada en amplicones se basa en fragmentos conservados y variables del gen 16S rRNA de bacterias y de la región ITS1 de hongos. Con la secuenciación del metagenoma completo se fragmenta y secuencia todo el ADN genómico presente en una muestra.
El uso de estos enfoques metagenómicos ha sido clave para caracterizar el desequilibrio de la composición microbiana en pacientes con DA, en comparación con sujetos sin DA84. La colonización de Staphylococcus aureus, una bacteria patógena que causa infecciones cutáneas, ha sido ampliamente reportada en la piel de la DA. El primer estudio longitudinal de muestras clínicas de DA pediátrica mediante secuenciación del ARNr 16S mostró grandes diferencias composicionales en la microbiota cutánea de los pacientes con DA frente a los controles (Kong et al., 2012). También se notificó una asociación entre la diversidad bacteriana y el tratamiento de la DA, así como una correlación entre el S. aureus y el S. epidermis y los brotes de la enfermedad. Estudios posteriores realizados con metagenómica de escopeta (del inglés, shotgun metagenomics) corroboraron estos hallazgos85. Aunque la colonización de S. aureus en la piel de la DA se observa de forma recurrente, aún no se ha desentrañado la contribución de este patógeno al inicio de la enfermedad.
El estudio de ratones modificados genéticamente ha permitido explorar el papel de la microbiota cutánea en el inicio de enfermedades inflamatorias de la piel. Se ha recapitulado la dermatitis eccematosa en ratones deficientes en los genes desintegrinas y ADAM17, que inducían disbiosis cutánea86. La piel de estos ratones estaba enriquecida en S. aureus, Corynebacterium mastiditis y Corynebacterium bovis. El tratamiento con antibióticos dirigidos contra estos patógenos revirtió el fenotipo de piel atópica y eliminó la inflamación, lo que demostró una probable relación causal entre las alteraciones del microbioma cutáneo y la dermatitis. Por lo tanto, se necesitan nuevas terapias dirigidas a S. aureus y comensales recurrentes enriquecidos en la piel de la DA para reemplazar los antimicrobianos de amplio espectro que se utilizan actualmente en estos pacientes.
En 2016, se analizó el perfil de la microbioma presente en la piel de muestras de voluntarios con DA, donantes sanos y personas con atopia no asociada a la DA (sujetos susceptibles a la DA), utilizando tanto la secuenciación del ARNr 16S como la secuenciación del metagenoma completo87. Se evaluaron tres métodos diferentes de muestreo de la piel (hisopado de la piel, cepillado con esponja y recogida con cinta adhesiva), concluyendo que la combinación de muestreo con cinta y secuenciación del metagenoma completo es un protocolo de análisis del microbioma de la piel fácil de usar y robusto. Se observaron que los géneros Streptococcus y Gemella eran más abundantes en la DA que en los donantes sanos, mientras que el Dermacoccus, el Deinococcus y el Methylobacterium eran más escasos en la DA. A nivel funcional, el microbioma de los sujetos con DA parecía estar preparado para producir un exceso de amoníaco, lo que explica los elevados niveles de pH que suelen observarse durante los brotes de dermatitis atópica. Además, se observó una mayor proporción de eucariotas en los individuos sanos que en la DA, así como un agotamiento de la familia Malasseziaceae en los individuos susceptibles a la DA. Se ha demostrado que distintos miembros del género Malassezia, que son hongos comensales asociados a la DA, inducen inflamación y exacerban los síntomas de la DA88.
De manera interesante, una disminución en la diversidad microbiana y la abundancia de gammaproteobacterias observadas en la piel de pacientes con DA y controles se han asociado a una menor biodiversidad ambiental en los alrededores de los hogares de los donantes de piel89. Se ha descubierto que las gammaproteobacterias, una clase asociada a la salud en la piel humana90, se correlacionan positivamente con los niveles de IL-10 en sangre, una citocina antiinflamatoria clave implicada en la tolerancia inmunológica.
En un estudio reciente, se recogió y se determinó el perfil del microbioma cutáneo de más de 822 muestras chinas y 538 muestras estadounidenses mediante metagenómica en escopeta, lo que dio lugar al catálogo integrado de genes microbianos de la piel humana (iHSMGC, por sus siglas en inglés)91. Este catálogo de especies bacterianas representa una herramienta de referencia muy valiosa para caracterizar la disbiosis cutánea asociada a la DA, permitiendo explorar la composición del resistoma de la DA, así como la función potencial de los comensales cutáneos en esta afección.
ConclusiónCualquiera de los enfoques moleculares descritos proporciona una gran cantidad de información nueva e interesante sobre la DA. Sin embargo, sólo una visión holística, clínicamente impulsada, que permita la integración de todas las “ómicas” hará posible con que toda esta información disponible conlleve a una medicina personalizada (fig. 4).
Para lograr este objetivo, es imprescindible aunar los esfuerzos de clínicos y especialistas en todas estas técnicas para obtener el mejor cuadro clínico -y el más realista- de los pacientes que vemos en nuestras consultas, las muestras más precisas en el momento adecuado, y las técnicas más innovadoras para poder liderar el salto cualitativo que suponen las posibilidades en “ómica”. Bajo esta premisa nace y se diseña el estudio SSAD, un estudio prospectivo multicéntrico que aglutinará el trabajo de más de 30 centros españoles especializados en DA.
FinanciaciónEste trabajo y los gastos de procesamiento del artículo han sido financiados por IMIDomics, FIS grant (PI21/01139) y proyecto EADV (PPRC 2021-22). Los patrocinadores no tuvieron ningún papel en el diseño, ejecución, interpretación o redacción del trabajo.
Conflicto de interesesSM es cofundador de IMIDomics, Inc. AJ es científico jefe de datos de IMIDomics, Inc. Los demás autores declaran no tener ningún conflicto de intereses.
Los autores agradecen a todos los médicos y profesionales sanitarios que han contribuido en el Consorcio Español IMID, así como a los Centros Clínicos AD participantes.